ThermoNet————点突变_3D-CNN预测
局限性:训练集受实验衍生的不稳定突变支配,并且机器学习方法易于过度拟合训练集
ThermoNet:一个基于深度3D-CNN的计算框架,用于预测单点突变时的∆∆Gs(ΔΔG为变化的吉布斯自由能)。 • 假设单点突变对蛋白质结构的整体结构产生的扰动可以忽略不计 • 条件:等位基因的3D结构是已确定的(实验确定或同源建模) 通过将蛋白质结构视为3D图像并对野生型结构和相应突变结构模型的突变位点周围的空间进行体素化来提取预测特征 每个体素用七个预定义的规则进行参数化 堆叠特征图以创建大小为[16,16,16,14]的张量作为十个深度3D-CNN的训练集的输入 每个分量3D-CNN模型由分别具有16、24和32个神经元的三个3D卷积层和一个24个神经元的密集连接层
体素7个性质值: GitHub存储库中提供了一个从突变列表创建输入张量的Python程序,网址为https://github.com/gersteinlab/ThermoNet。
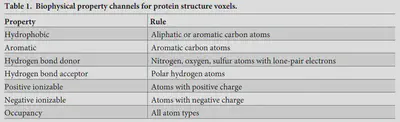
总结:首先获得蛋白质的三维结构,然后经过Rosetta处理分别得到细化后的野生型结构和点突变结构,将突变结构处以突变点为中心添加16埃的3D体素网格,并根据七个理化性质(疏水性、芳香性、氢键给体、氢键受体、正离子、负离子、占有)得到七个161616的体素网格,将七个组合成为[16,16,16,7]的特征图,点突变结构同理。堆叠两个161616*7的特征图得到[16,16,16,14]张量。将其用作卷积神经网络的输入。卷积层有三层,分别包含16,24,32个神经元。压平卷积层的输出,使其转化为密集连接层的输入,经过24层的密集连接层输出由点突变引起的相对野生型结构预测的ΔΔG。实验参数通过五层交叉验证调整。
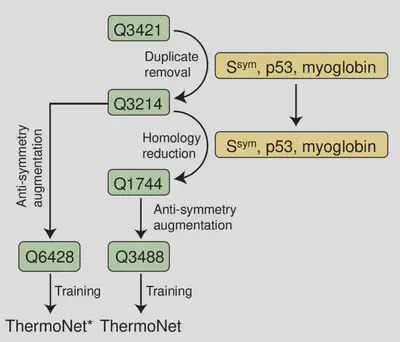
数据集问题:数据泄露——训练集中包含与测试集中同源蛋白质,会出现数据泄露。(mCSM和INPS解决) ThermoNet解决数据集问题:删除重复的数据点和裁剪蛋白质同源性水平(∆∆G的非对称性质扩增数据) 数据集地址:https://github.com/gersteinlab/ThermoNet
更稳定的蛋白质,意味着更高比例的蛋白质处于折叠形式,具有更负的∆G值。
参考文献: Li B, Yang YT, Capra JA, Gerstein MB. Predicting changes in protein thermodynamic stability upon point mutation with deep 3D convolutional neural networks. PLoS Comput Biol. 2020 Nov 30;16(11):e1008291. doi: 10.1371/journal.pcbi.1008291. PMID: 33253214; PMCID: PMC7728386.